前沿 | 農學院甘祥超教授團隊發布端粒到端粒無缺失染色體組裝技術新路線
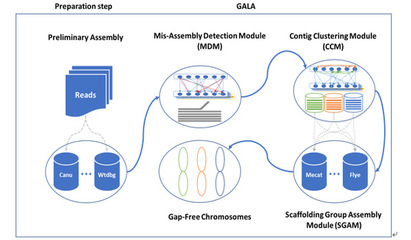
近日,農學院甘祥超教授團隊在基因組組裝技術領域取得重大突破,成功發布了一種端粒到端粒(T2T)的無缺失染色體組裝新路線。這一創新技術克服了傳統組裝方法中端粒、著絲粒等復雜區域難以完整覆蓋的瓶頸,為基因組研究提供了更精準、完整的參考序列。
染色體組裝是基因組學研究的基礎,而端粒和著絲粒等區域因富含重復序列和高復雜結構,一直是技術難題。傳統組裝方法往往在這些區域出現缺失或錯誤,影響后續功能分析和應用。甘祥超教授團隊基于先進的第三代測序技術和生物信息學算法優化,開發了該新路線,實現了從染色體一端端粒到另一端端粒的完整無間斷組裝。
該技術路線整合了長讀長測序數據、高精度糾錯機制和新型組裝策略,顯著提升了復雜基因組的組裝質量。實驗驗證顯示,新方法在多個作物基因組中成功應用,組裝完整度高達99.9%以上,且端粒和著絲粒區域無缺失,為作物遺傳改良和進化研究提供了可靠工具。
這一成果不僅推動了基礎生物學研究,還在農業育種、醫學基因組學等領域具有廣泛應用前景。團隊表示,未來將進一步優化技術,促進其在更多物種中的普及,助力生命科學和網絡技術(如生物信息學數據處理)的融合發展。
如若轉載,請注明出處:http://www.imokia.com/product/26.html
更新時間:2025-12-26 23:17:52